ChatGPT 推出AI圖像生成功能:免費用戶每日限用三次
421
在生物序列建模领域,深度学习技术的进步令人瞩目,但高昂的计算需求和对大数据集的依赖让许多研究者感到困扰。最近,麻省理工学院(MIT)、哈佛大学和卡内基梅隆大学的研究团队推出了一种名为 Lyra 的新型生物序列建模方法。这种方法不仅参数显著减少到仅有传统模型的12万分之一,而且能够在短短两小时内使用两块 GPU 进行训练,极大地提升了模型的效率。
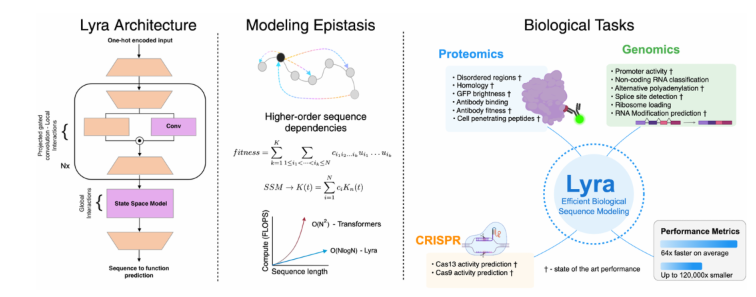
Lyra 的设计灵感来源于生物学中的上位效应(即序列内突变间的相互作用),它通过一个次二次架构来有效理解生物序列与其功能之间的关系。这种新模型在100多个生物任务中展现出色的性能,包括蛋白质适应度预测、RNA 功能分析及 CRISPR 设计等领域,甚至在某些关键应用中达到了当前技术的最佳性能(SOTA)。
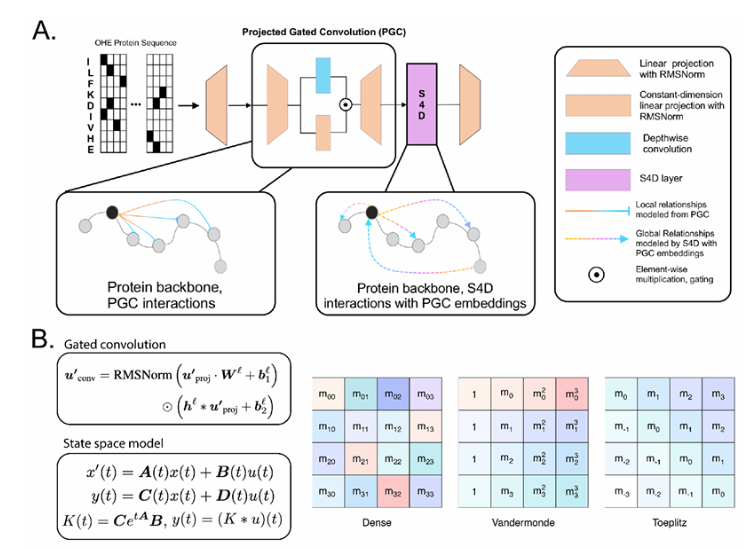
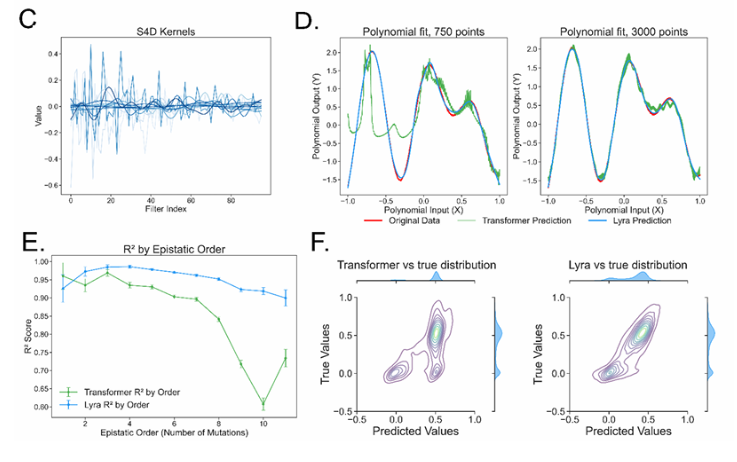
与传统的卷积神经网络(CNN)和 Transformer 模型相比,Lyra 的推理速度提升了64.18倍,同时大幅降低了参数需求。这得益于其创新的混合模型结构,Lyra 结合了状态空间模型(SSM)和投影门控卷积(PGC)来捕获生物序列中的局部和全局依赖关系。SSM 通过快速傅里叶变换(FFT)高效建模全局关系,而 PGC 则专注于提取局部特征,二者的结合让 Lyra 在计算效率和可解释性之间达成了良好平衡。
Lyra 的高效性不仅能够推动基础生物研究的进展,也可能在治疗开发、病原体监测以及生物制造等实际应用中发挥重要作用。研究团队希望,通过 Lyra,更多的研究者能够在资源有限的情况下进行复杂的生物序列建模,从而加速生物科学的探索。